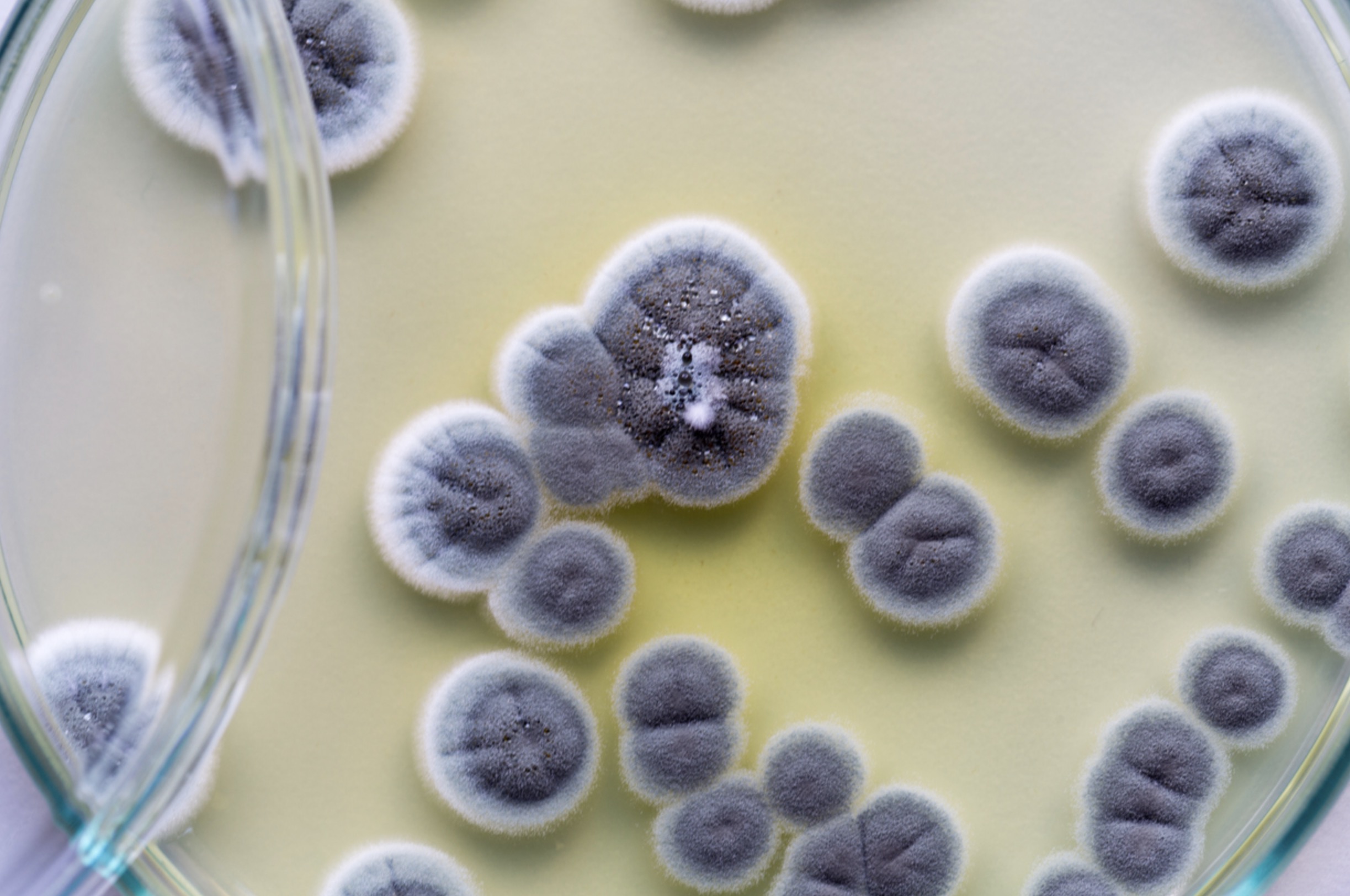
La resistencia a los antifúngicos es una amenaza creciente para la salud global y ha sido recientemente incorporada a la lista de prioridades sanitarias de la Organización Mundial de la Salud (OMS). Sin embargo, los hongos han recibido menos atención que las bacterias en los programas de vigilancia y desarrollo de fármacos, a pesar de su impacto clínico y de provocar pérdidas importantes en cultivos agrícolas.
Igualmente, aunque el auge de la secuenciación masiva de ADN ha permitido lograr grandes avances en la investigación del microbioma y la resistencia a antibióticos, la falta de bases de datos exhaustivas, la ausencia de referencias y la complejidad del genoma eucariótico hace que la investigación de la resistencia a antifúngicos, desde un punto de vista in silico y a gran escala, sea un reto a superar.
Para hacer frente a este desafío, un equipo internacional liderado por el Instituto de Biología Funcional y Genómica (IBFG, CSIC–USAL), la Universidad de Salamanca y la Université Laval (Canadá), junto con el apoyo de distintas instituciones de Canadá y Países Bajos, ha creado FungAMR, una base de datos que recopila más de 50.000 entradas y 35.000 mutaciones genéticas identificadas en 95 especies de hongos de interés clínico, agrícola y ambiental, asociadas a 246 proteínas y que confieren resistencia a un total de 208 antifúngicos. Esta información ha sido extraída manualmente de más de 500 estudios científicos y clasificada rigurosamente según el nivel de evidencia experimental, lo que permite a los usuarios evaluar la fiabilidad y el respaldo científico de cada mutación. El trabajo ha sido publicado recientemente en la revista Nature Microbiology.
“La motivación de FungAMR residía en la imperiosa necesidad de poder tener una base de datos exhaustiva, detallada y fiable sobre mecanismos de resistencia a antifúngicos. Poder disponer de FungAMR en una interfaz web amigable e intuitiva va a facilitar mucho la utilidad por parte del usuario”, destaca Christian R. Landry, investigador de la Université Laval.
Además, el equipo ha desarrollado el software ChroQueTas (Chromosome Query Targets), una herramienta bionformática pionera que permite analizar genomas y/o proteomas fúngicos y detectar automáticamente mutaciones que confieren resistencia a antifúngicos. Disponible como software libre, ChroQueTas facilita el cribado genético de cepas clínicas y ambientales de forma rápida, precisa y escalable.
“En las pruebas que realizamos con genomas de estudios publicados, ChroQueTas ha reportado las mutaciones descritas con anterioridad de manera fiable, así como ha identificado mutaciones no reportadas previamente en esos genomas, lo que demuestra su potencial como instrumento clave para la vigilancia genómica”, señala Narciso M. Quijada, investigador del Instituto de Biología Funcional y Genómica (IBFG)
El trabajo revela que muchas mutaciones confieren resistencia a múltiples antifúngicos a la vez (fenómeno de resistencia cruzada), como consecuencia de su forma de actuación similar, y que esta resistencia puede surgir a partir del mismo mecanismo en especies distintas. El uso masivo de antifúngicos en agricultura —especialmente azoles— se identifica como una de las principales presiones evolutivas que impulsan este problema. Esta situación dificulta el tratamiento de infecciones fúngicas y subraya la necesidad urgente de desarrollar nuevas terapias con mecanismos de acción alternativos.