En el proceso de transcripción del ADN en proteínas es clave conocer la forma en que esta se realiza, así como los factores que pueden influir en su regulación. Desde hace tiempo se sabe que existen modificaciones químicas que condicionan el mecanismo y que pueden afectar no solo al ADN, sino también al ARN, la molécula que permite que la información genética sea comprendida por las células para formar proteínas. Un equipo de investigadores del Consejo Superior de Investigaciones Científicas (CSIC) ha hecho un análisis de los avances en el estudio de las modificaciones químicas más relevantes del ARN, también denominadas marcas epitranscriptómicas. Si se logra revertir estas marcas, que en el cáncer pueden estar mal colocadas, sería posible controlar la expresión de los genes, o la actividad de determinadas proteínas y, por tanto, proporcionarían el desarrollo de nuevas dianas farmacológicas contra la enfermedad. La revisión ha sido publicada en la revista Molecular Cancer por investigadores del Centro de Investigación del Cáncer (CIC-IBMCC), mixto de la Universidad de Salamanca y el CSIC.
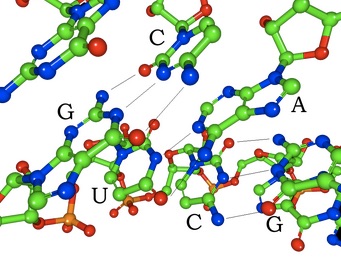
Aunque se sabe que existen más de 170 modificaciones del ARN (cuyo conjunto se conoce como epitranscriptoma), su papel biológico sigue siendo muy desconocido. En la última década, los investigadores han avanzado en el conocimiento de sus funciones y se ha descubierto que el exceso o la falta de algunas de ellas condicionan el progreso de diferentes tipos de cáncer.
Esta revisión se centra en las denominadas modificaciones postranscripcionales, errores cuya acumulación de forma aberrante se relaciona con la supervivencia, la metástasis y la resistencia a las terapias, características clave en el desarrollo y progresión del cáncer. “Se trata de moléculas químicas que se pegan a los componentes principales del ARN (las bases nitrogenadas) por unas proteínas especializadas de la célula, las enzimas modificadoras de ARN. Cuando estas enzimas se expresan más o menos de lo que deberían o su actividad está activada o reprimida por mutaciones, se producen enfermedades, como el cáncer o patologías neurológicas”, indica Sandra Blanco, investigadora del Centro de Investigación del Cáncer (CIC-IBMCC) y autora del trabajo.
En concreto, en el artículo se analizan tres modificaciones: la metilación de adenina-6, la metilación de citosina-5 y la pseudouridina. Saber si el código epitranscriptómico en un tumor en concreto está alterado, por ejemplo, cuando hay más metilación adenina-6 de la que debería en las células, permitiría el diseño de moléculas que la repriman. “Si se reprime esa metilación que necesitan en exceso las células tumorales para seguir creciendo, éstas morirán. Además, se trata de un mecanismo que no afecta a un solo tipo de tumor, sino que puede observarse en distintas patologías como gliomas, leucemias, mielomas o cáncer de mama y pulmón, entre otros”, explica Blanco.
Otras modificaciones, como la metilación citosina-5, son necesarias para que las células cancerígenas sobrevivan a las señales de estrés, por lo que, al reprimir esa modificación usando moléculas inhibidoras de la metilación, las células tumorales se vuelven más sensibles a los tratamientos quimioterapéuticos.
Los científicos esperan combinar los dos tipos de fármacos para diseñar tratamientos más eficaces. “Hasta la fecha no existen o no se han diseñado fármacos que inhiban la formación de las modificaciones del ARN. Esto es debido a que es un campo de investigación muy nuevo. Solo en los tres últimos años hemos descubierto, por ejemplo, la implicación de la epitranscriptoma en el cáncer, y apenas hemos estudiado algunas de esas modificaciones. El siguiente paso será diseñar esos fármacos que nos permitan reescribir el código epitranscriptómico en una célula tumoral. En un futuro encaminado a la medicina personalizada, este tipo de marcadores podrían ayudar a encontrar el mejor tratamiento dependiendo de las modificaciones y características de un paciente en concreto”, concluye.
CIC-IBMCC Comunicación / CSIC Comunicación